espace pédagogique > disciplines du second degré > svt > enseignement > lycée > Spécialité SVT > Première > Le fonctionnement du système immunitaire humain
immunologie en terminale S : utiliser les données numériques et les logiciels lors des activités pratiques
mis à jour le 16/12/2005
Un ensemble d'activités pratiques pouvant être mises en oeuvre pour traiter le programme d'immunologie en Terminale S. Les logiciels mesurim, anagène, rastop et winMDI sont utilisés.
mots clés : logiciels, immunité, lymphocytes, VIH, anticorps, WinMDI, mesurim, rastop, molécules, antigène, infection, SIDA, Anagene, Tice, tutoriel
Extraction de travaux de différentes équipes INRP et de Jean-François MADRE (Académie d'Amiens).
Sommaire
Objectifs
Quantifier un electrophorégramme avec Mesurim_pro
Etudier un anticorps avec Rastop
Etudier la formation des complexes immuns avec Rastop
Comparer différents anticorps avec Rastop et Anagène
Mesurer les différentes populations cellulaires avec
WinMDI
Le chapitre d'immunologie du programme de terminale S est l'occasion de mettre les élèves en situation de réaliser techniquement, en leur proposant des manipulations variées et motivantes, de la réalisation de frottis sanguins (non humains) à la réalisation de test immunologiques. Ces séances de TP sont fondamentales et on ne peut s'en dispenser. Elles peuvent être complétées de façon intéressante par des activités utilisant les fonctionnalités de quelques logiciels comme Anagène, Rastop, WinMDI et Mesurim.
Quantifier un électrophorégramme
avec Mesurim_pro
Le logiciel Mesurim Pro de Jean-François Madre est téléchargeable gratuitement sur le site de l'académie d'Amiens. Après l'installation une icône permet d'ouvrir le logiciel. |
http://svt.ac-amiens.fr/archives_svt/info/logiciels/Mesurim2/Index.htm
|
L'électrophorégramme est une technique élémentaire permettant de mettre en évidence l'augmentation de la quantité d'anticorps plasmatiques. Le logiciel mesurim_pro comporte dans son stock d'images une image numérisée d'une bande d'électrophorèse. Le fichier s'appelle plasma.jpg Les commandes "Fichier" puis "Ouvrir" permettent d'afficher l'image. On voit clairement que le dépôt s'est effectué à droite et que la migration a eu lieu de droite à gauche. Cela correspond à la plupart des diagrammes présentés dans les manuels et nous allons garder cette disposition. Un travail sur la signification des différentes taches peut déjà être fait par les élèves. |
 |
Choisir l'outil "mesure" et tracer un trait selon la direction choisie sur le cliché. |
 |
Dans la liste déroulante des types de mesure, choisir "Lumière sur une bande". Dans la boîte de paramétrage qui s'ouvre alors, changer la largeur de la bande et choisir "Mesure en absorption". |
 |
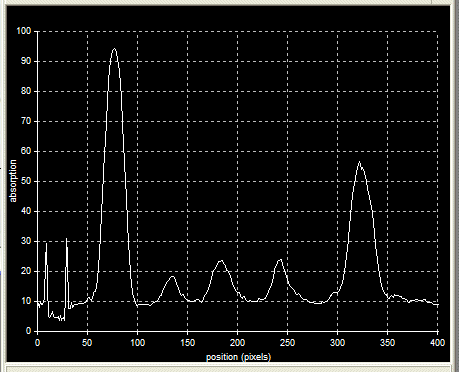
| Le diagramme d'absorption de la lumière par l'électrophorégramme montre plusieurs pics caractéristiques. De gauche à droite les albumines puis les globulines alpha1, alpha2, beta et gamma. |  |
| En utilisant des électrophorégrammes pathologiques, on peut montrer que la présence d'un antigène (ici les cellules cancéreuses d'un myélome) provoque une augmentation globale de la production des IgG. |  |
Ici, il s'agit d'une cirrhose du foie.
Il serait très intéressant de posséder une bande d'électrophorèse relative à un épisode grippal ou à une hépatite pour montrer quel type de réaction provoque l'introduction d'un virus dans l'organisme. |
 |
Etudier un anticorps
avec Rastop
| Rastop 2.03vf est la dernière version mise à la disposition des enseignants gratuitement par l'INRP. | http://www.inrp.fr/Acces/biotic/rastop/accueil.htm Voir également les travaux sur Rastop sur le site de l'INRP |
| La seule condition est de s'enregistrer auprès
du serveur et d'avoir une connexion assez rapide. Décompactez le
fichier directement sur l'unité disque C, par exemple. Un répertoire rastopvf est créé automatiquement mais il n'y a pas de procédure d'installation proprement dite. Il suffit donc de créer un raccourci sur le bureau pour le programme "Rastop.exe" |
 |
Rastop utilise des fichiers au format .pdb (protein data bank). Pour les données de base, le plus simple est d'utiliser les données de Rasmol mais il est aussi possible d'utiliser les fichiers mis à disposition par l'INRP ou les serveurs de données .pdb. Dans ce cas, le plus simple est de faire la requête <nom de la molécule> + .pdb dans le moteur de recherche de votre choix. |
|
| Le fichier de molécule IGG-TOTAL.pdb correspondant à un d'anticorps circulant anti-lysozyme fait partie des fichiers de la banque de données initiale de Rasmol. | |
Il suffit de choisir le fichier IGGTOTAL.pdb pour voir apparaître la forme globale de la molécule. Le mode d'affichage par défaut montre uniquement
les liaisons. Pour se rapprocher des représentation usuelles en
chimie, il est préférable d'afficher en sphères de
Van der Waals avec le bouton : Le résultat montre que chaque couleur correspond à un élément.
Le Carbone est en gris, l'Oxygène en rouge, l'Azote en bleu et le Souffre en jaune. On notera que l'Hydrogène n'est pas représenté. |
 |
| La première approche de la molécule se fera par la mise en évidence des chaînes lourdes et légères.
Les chaines légères sont ici en jaune et en vert tandis que les chaînes colorées en bleu correspondent aux chaînes lourdes. La partie en rouge correspond à un glucide qui n'entre pas dans le cadre de l'étude des anticorps en Terminale S. |
 |
Pour répondre à la question des liaisons qui assurent la cohésion des chaînes, il est nécessaire d'adopter un autre mode de représentation en utilisant les connaissances de première sur la structure des protéines. On fera donc successivement afficher le squelette carboné,
puis effacer les atomes :
|
On remarquera que le glucide est effacé mais que la nature des liaisons entre les chaînes reste mystérieuse. |
L'affichage des ponts disulfures répond au problème posé mais la notion de pont disulfure est difficile à expliquer.
|
Le logiciel affiche les ponts disulfure avec beaucoup trop de discrétion. |
Pour les mettre en évidence il est possible
de les colorer spécifiquement. L'utilisation du bouton Choisir "Ponts disulfures" dans la liste déroulante puis une couleur. Ils sont un peu plus visibles. |
 |
La sélection des seuls atomes de souffre se fait dans la liste déroulante la plus à gauche. Une fois l'élément choisi, la sélection n'est effective
qu'en actionnant le bouton |
 |
Les atomes de souffre seront alors affichés
en utilisant l'icône "Boules et Bâtonnets" la sélection d'une couleur pour les atomes dans la palette termine la mise en évidence des ponts disulfures.
|
 |
A partir de cet affichage, il est possible de produire un schéma de la molécule d'anticorps avant de passer à l'étude de l'interaction avec les antigènes. |
 |
Etudier la formation des complexes immuns avec Rastop
Le fichier IGG-LYS.PDB contient la représentation des extrémités des chaînes lourdes et légères (Le fragment FAB) associées à l'antigène constitué par une molécule de lysozyme.
|
 |
| En utilisant l'affichage en sphère VDW puis la coloration par chaîne
il est possible de distinguer l'antigène en rouge, l'extrémité de la chaîne lourde en bleu et celle de la chaîne légère en vert. |
 |
| A ce stade, l'hypothèse d'une complémentarité géométrique peut être émise. Pour la vérifier nous allons utiliser les fonctions de zoom et de coupe virtuelle. | |
| En sélectionnant Trans/Zoom, les actions sur les curseurs sont interprétées comme des translations selon l'un des trois axes. Cela permet de centrer l'observation sur la zone de contact entre anticoprs et antigène. |  |
La fonction de coupe est commandée par un petit tableau situé en centre de la barre inférieure.
L'action sur le bouton de droite déplace un plan de coupe virtuel vers l'arrière de l'écran. Cela permet de constater qu'il existe une complémentarité géométrique en trois dimensions entre antigène et anticorps. |
 |
Comparer différents anticorps
avec Rastop et Anagène
L'étude des western blot comme moyens de dépistage de la séropositivité pose le problème de la diversité des anticorps fabriqués par l'organisme infecté par le VIH. Les anticorps se lient spécifiquement à l'une des protéines de la bande. Une hypothèse basée sur l'analogie avec l'interaction enzyme substrat vue en Première S peut être formulée par les élèves. Il s'agirait de différences dans les séquences peptidiques des anticorps. Il est possible de poser le problème en affichant aussi deux vues de fragments FAB de complexes immuns formés avec différentes protéines du VIH. |
Ces ressources sont disponibles sur le site de l'INRP à l'adresse : http://www.inrp.fr/Acces/biotic/immuno/html/telechar.htm Il faut ensuite télécharger le fichier IGG-SIDA-3D.zip pour rastop et igg-vih-8seq.edi pour les données "Anagène" correspondantes. Le fichier Zip sera décompressé dans le dossier adéquat. 8 fichiers au format pdb présentent des fragments FAB soit seuls soit associés à un antigène du VIH. |
Ouvrir une nouvelle fenêtre dans rastop.
Ouvrir dans cette fenêtre le fichier ACY_gp120.pdb
En utilisant le bouton On voit ici que le fragement FAB correspond à un anticorps associé à la GP120 |
|
Pour avoir des informations sur la molécule utiliser la commande :
|
|
 |
|
|
Faire à nouveau les commandes pour une nouvelle fenêtre et l'ouverture d'un nouveau fichier. 1E6J correspond à un anticorps contre la P24. Le bouton En cliquant sur chaque partie de la molécule, il est possible d'afficher le nom de la chaîne pointée
Il s'agit ici de la chaîne H qui colorée en bleu alors que la chaîne légère (L) est en vert et que l'antigène est en rouge (P) |
Le logiciel anagène distribué par l'INRP va servir à vérifier la validité de l'hypothèse formulée au début de l'activité. Il permet de comparer les séquences de différentes protéines. Les séquences sont dans le fichier igg-vih-8seq.edi
Toutes les chaînes sont affichées sans considération d'alignement. |
|
 |
|
Pour que les bases de la comparaison soient valides il convient de
ne comparer que des chaînes de même type. A l'aide de la souris
il faut mettre les quatre chaînes H en surbrillance puis utiliser
l'outil de comparaison
|
 |
La première ligne de la fenêtre de comparaison indique par des étoiles les identités totales et par des points les identités partielles. Cliquer sur la règle pour afficher les numéros des acides aminés. On remarque qu'au début des séquences ces identités sont très rares. |
|
| Les identités deviennent très importantes à partir de l'acide aminé 118 pour la chaîne lourde. Les élèves noteront que la partie variable des fragments FAB se trouvent entre 1 et 118 pour la chaîne lourde. Il est possible à ce stade de faire mettre en évidence des parties hypervariables où les identités sont inexistantes. On notera les numéros de la même façon. On fera ce travail aussi sur les chaînes légères. |
|
Pour localiser les zones variables des fragments FAB dans rastop nous
allons changer le mode de représentation des protéines de
l'anticorps. Pour sélectionner la chaîne lourde, utiliser
le bouton
|
 |
La fenêtre inférieure indique le nombre d'atomes sélectionnés. Pour afficher la partie sélectionnée en ruban, utiliser la commande spécifique. Cela ne suffit pas car il faut aussi faire disparaître l'affichage des atomes.
|
 |
|
La chaîne lourde apparaît en squelette carboné. Faire de même pour la chaîne légère et utilisant cette fois *L.
Pour sélectionner la zone variable, l'expression change. Il faut faire attention à laisser des espaces entre les différentes parties de l'expression.
Il s'agit ici de sélectionner les acides aminés 1 à 112 de la chaîne légère. |
Une fois la sélection réalisée, on utilisera
la représentation en sphère Ici les 112 premiers acides aminés de la chaîne légère sont effectivement ceux qui sont les plus proches de l'antigène. Le même travail sera réalisé sur la chaîne lourde. |
 |
Si le travail porte sur les parties hypervariables, la proximité entre ces atomes et ceux de l'antigène est évidente. Le problème de l'origine de la variabilité des anticorps pourra alors être posé. |
|
Mesurer les différentes
populations cellulaires avec WinMDI
Il est classique de faire utiliser une courbe montrant l'évolution des populations de lymphocytes au cours de l'infection par le VIH. La question de techniques qui permettent les comptages est souvent posée par les élèves. Les manipulations proposées ci-dessous utilisent WinMDI, un logiciel pemettant de traiter les résultats de comptages réalisés par cytométrie de flux. Les cellules extraites d'une prise de sang sont marquées par des substances fluorescentes liées à des anticorps dirigés contre les molécules CD. A chaque molécule anti-CD est liée une substance fluorescente de couleur différente. Les cellules sont alors amenées à circuler dans un tube très fin qui n'en laisse passer qu'une seule à la fois. Elles passent devant des stimulateurs et des détecteurs de fluorescence spécifiques des longeurs d'ondes émises. Chaque passage de cellule est détecté par la fluorescence émise, incrémente un compteur et enregistre le niveau de fluorescence correspondant à la cellule. |
Le logiciel WinMDI est téléchargeable sur le site : Les données cytométriques correspondent
au fichier http://www.inrp.fr/Acces/biotic/immuno/html/telechar.htm Des informations supplémentaires sur les fichiers sont disponibles sur la page : http://www.inrp.fr/Acces/biotic/immuno/html/ressourc.htm Voir aussi les pages spécifiques sur le serveur de l''INRP
|
L'installation du logiciel et des données se fait de façon classique . Un clic sur l'icône provoque l'ouverture du logiciel. |
 |
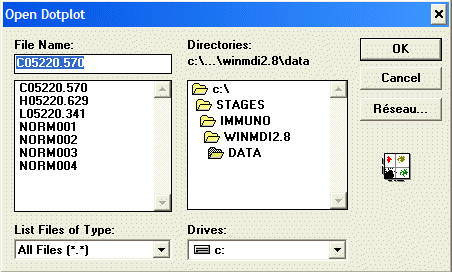
| Une fenêtre propose le type de représentation à utiliser. Il faut choisir dotplot pour pouvoir caractériser les lymphocytes T4 ou T8 par deux paramètres cytométriques. |  |
Le fichier C05220.570 correspond à une personne non infectée. Les fichiers L05220.341 à un malade asymptomatique et H05220.629 à un SIDA déclaré. |
 |
Le paramétrage du diagramme est la phase la plus délicate. Il repose sur quelques connaissances essentielles concernant les marqueurs membranaires des lymphocytes : Tous les lymphocytes possèdent le marqueur CD3, Il faut donc choisir comme variable X les CD3 et comme Y les CD4. Il faut aussi cocher les cases selon le modèle ci-contre. |
 |
En cliquant sur OK, on voit apparaître un diagramme où chaque point représente une cellule. On voit nettement que plusieurs populations cellulaires sont présentes dans l'échantillon. Les mesures de fluorescences sont telles que toute fluorescence inférieure à 10² est considérée comme non significative. Autrement dit, seules les cellules présentant une fluorescence supérieure à 10² pour les CD3 et CD4 pourront être considérées comme des lymphocytes T4. Nous voyons effectivement une population présentant ces caractéristiques. Une approche semi quantitative est maintenant possible. |
 |
Pour ouvrir une seconde fenêtre
il faut utiliser la commande |
|
On voit ici successivement les diagrammes des sujets sain, infecté asymptomatique et malade. La population des lymphocytes T4 a fortement diminué chez ce dernier. |
|
Il s'agit maintenant de dénombrer les lymphocytes T4. En cliquant avec le bouton gauche de la souris sur le graphique, on affiche un menu contextuel dans lequel il faut choisir "Regions".
|
 |
La boîte de dialogue permet de créer une première région. Le pointeur adopte la forme d'une croix et permet de définir un polygone par des clics successifs.
|
 |
Il est important de délimiter au plus près le groupe de cellules à mesurer. Un petit panneau de contrôle permet de valider la sélection.
|
 |
Pour afficher la sélection, revenir au menu contextuel et choisir "Stats" En consultant les valeurs numériques on constate que les lymphocytes T4 représentent 928 cellules dans l'échantillon de sang. Il est préférable de ne pas utiliser les pourcentages car le nombre total de Lymphocytes T n'est pas stable dans le temps dans une infection par le VIH. Le même travail devra être entrepris sur les deux autres diagrammes. |
 |
Le paramètre utilisé par les médecins n'est pas le nombre de T4 mais le rapport T4/T8. Il faut donc modifier la variable affichée en Y pour pouvoir mettre en évidence les cellules porteuses de CD8. Cela se fait par le menu contextuel en activant "Format".
|
Il faut alors simplement modifier la variable Y. En utilisant le protocole ci-dessus, il sera ainsi possible de dénombrer les T8. On fera les mêmes manipulations pour les deux autres diagrammes. L'évolution du rapport T4/T8 pourra ainsi être étudiée.
|
Travail mis en page pour son utilisation dans une session
de préparation à l'agrégation interne de SVT par
François
CORDELLIER, Professeur de SVT au lycée Jean Perrin de Rezé
Monique Dupuis, IPR-IA de SVT dans l'académie de Nantes
information(s) pédagogique(s)
niveau : 1ère
type pédagogique : travaux pratiques, démarche pédagogique
public visé : enseignant
contexte d'usage : atelier, classe, espace documentaire, laboratoire, salle multimedia
référence aux programmes :
Immunologie
L'immunité adaptative, prolongement de l'immunité innée
fichier joint
information(s) technique(s) : fichier pdf
taille : 737 ko ;
sciences de la vie et de la Terre - Rectorat de l'Académie de Nantes

 s'identifier
s'identifier
 portail personnel ETNA
portail personnel ETNA
























 .
Cela permet d'ouvrir un second fichier avec les mêmes caractéristiques
de représentation que la première. Il faudra faire de même
pour le troisième fichier.
.
Cela permet d'ouvrir un second fichier avec les mêmes caractéristiques
de représentation que la première. Il faudra faire de même
pour le troisième fichier.



